Wissenschaftler visualisieren biochemische Prozesse
Ein Bild sagt mehr als tausend Worte
Kontinuierlich weiterentwickelte Computerprogramme ermöglichen es, immer komplexere biochemische Vorgänge zu simulieren – mit dem Ergebnis, dass die auszuwertenden zeitabhängigen Datensätze Dateigrößen von mehreren Gigabyte erreichen können und bis zu einer Million Partikel pro Zeitschritt enthalten. Um Simulationsdaten in dieser Größenordnung und Komplexität interaktiv analysieren zu können, entwickeln Wissenschaftler des Sonderforschungsbereichs (SFB) 716 und des Exzellenzclusters Simulation Technology (SimTech) am Visualisierungsinstitut der Universität Stuttgart (VISUS) derzeit Visualisierungstechniken. Diese sollen es dem Benutzer erlauben, die immense Datenflut zu erfassen.
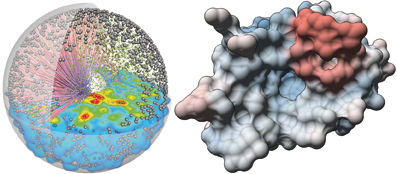
Visualisiertes Zellmodell (links) zeigt Transportprozesse von Signalproteinen. Rechts: Oberflächendarstellung eines Proteins mit eingefärbtem Temperaturfaktor.
VISUS
„Ein Bild sagt mehr als tausend Worte“, heißt ein berühmtes Sprichwort. Auch für Wissenschaftler ist die Visualisierung von Simulationsdaten enorm wichtig, um sie besser analysieren zu können. Bei der Visualisierung der zunehmend komplexer und umfangreicher werdenden Simulationen, beispielsweise von Konformationsänderungen in Proteinen oder von enzymatischen Reaktionen, stoßen die Visualisierer an Grenzen. Der Grund: die Speicherbandbreite entwickelt sich zu einem Flaschenhals und die Rechenzeit steigt in Abhängigkeit von der Datensatzgröße an. Dieser Herausforderung haben sich die
VISUS-Mitarbeiter gestellt: Für zwei Teilprojekte innerhalb des SFB 716 und SimTech mit jeweils verschiedenen Zielsetzungen in der Anwendung konnten sie ähnliche Visualisierungstechniken für Daten mit unterschiedlichem Skalen- und Abstraktionsgrad entwickeln. In beiden Projekten haben die Stuttgarter Wissenschaftler mit partikelbasierten Daten gearbeitet, wobei ein Partikel in der Simulation für ein reales Atom oder Molekül steht. Um eine interaktive Analyse, Simulation und Visualisierung zu gewährleisten, haben die Forscher parallele Algorithmen entwickelt, welche die Fähigkeiten aktueller Mehrkernprozessoren und programmierbarer Grafikkarten ausnutzen, da mehrere Berechnungen gleichzeitig ausgeführt werden können.
Im Sonderforschungsbereich 716 untersuchen die Forscher mit Hilfe von Molekulardynamiksimulationen unter anderem, wie sich Proteinmoleküle auf atomarer Ebene in unterschiedlichen Lösungsmitteln verhalten. Beispielsweise analysieren die Wissenschaftler, wie das Mischungsverhältnis sein muss, damit Proteine mehr Biokraftstoff synthetisieren. Durch das am VISUS entwickelte interaktive Visualisierungsprogramm MegaMol können die Forscher die Simulationsergebnisse visualisieren und das dynamische Verhalten der Moleküle als Animation sehen. Darüber hinaus kann der Benutzer beispielsweise die Daten von beliebigen Blickwinkeln betrachten und zwischen verschiedenen Einfärbungen wählen, um unterschiedliche Merkmale sichtbar zu machen.
Im Rahmen von SimTech simulieren die Forscher mittels Monte-Carlo-Simulationsmethoden beispielsweise Transportprozesse von Signalen in Zellen, um möglicherweise neue Ansatzpunkte für die Krebstherapie zu finden. Dabei werden aus vielen Atomen bestehende Moleküle durch einzelne Partikel abstrahiert. Diese Vereinfachung erlaubt es, Vorgänge zu simulieren, die in der Realität mehrere Minuten in Anspruch nehmen, während mit Molekulardynamiksimulationen nur Vorgänge im Bereich von Nanosekunden untersucht werden können. Für die Analyse der Simulationsdaten spielt das entwickelte Visualisierungswerkzeug CellVis eine große Rolle.
Meistgelesene News
Organisationen
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Chemie-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.




























































