Neue, offene Software für hochauflösende Mikroskopie
Mit ihren Spezialmikroskopen können Experimentalphysiker bereits einzelne Moleküle beobachten. Im Gegensatz zu herkömmlichen Lichtmikroskopen müssen die rohen Bilddaten mancher ultrahochauflösender Geräte aber erst bearbeitet werden, damit ein Bild entsteht. Für die ultrahochauflösende Fluoreszenzmikroskopie, die auch an der Universität Bielefeld in der biophysikalischen Forschung zum Einsatz kommt, haben nun Mitarbeiter der Arbeitsgruppe Biomolekulare Photonik eine neue, offene Softwarelösung entwickelt, um Rohdaten schnell und effizient bearbeiten zu können. Der Bielefelder Physiker Dr. Marcel Müller berichtet in der neuesten Ausgabe der Zeitschrift „Nature Communications“ über die frei zugängliche, neue Software.
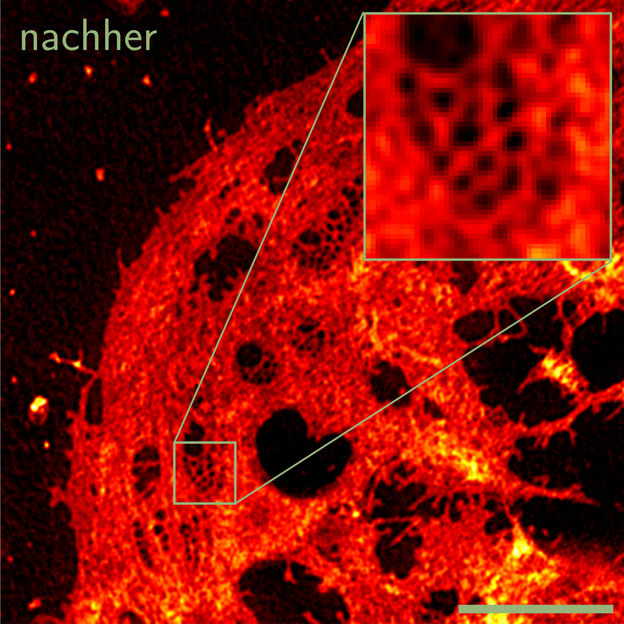
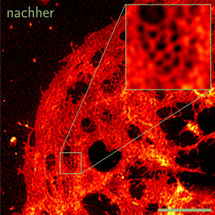
Die Aufnahme zeigt eine Leberzelle nach der Auswertung der Daten durch die an der Universität Bielefeld entwickelten Software.
Universität Bielefeld
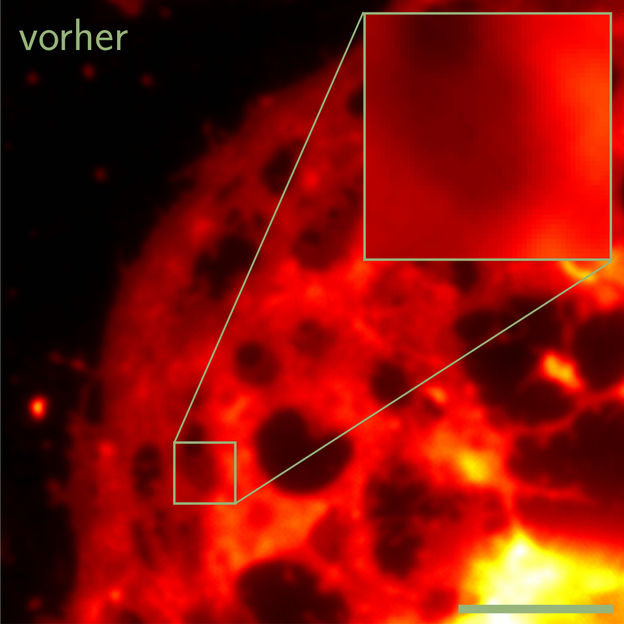
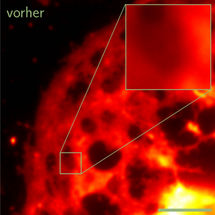
Die Aufnahme zeigt eine Leberzelle vor der Auswertung der Daten durch die an der Universität Bielefeld entwickelten Software.
Universität Bielefeld
Herkömmliche Lichtmikroskopie kann nur eine definierte untere Auflösungsgrenze erreichen, die durch die Beugung des Lichts auf circa 1/4 eines Mikrometers beschränkt ist. Die hochauflösende Fluoreszenzmikroskopie ermöglicht es, Bilder mit einer Auflösung deutlich unter dieser physikalischen Grenze zu erhalten. Für die Entwicklung dieser für die biomedizinische Forschung wichtigen Schlüsseltechnologie wurden die Physiker Stefan Hell, Eric Betzig und William Moerner 2014 mit dem Nobelpreis ausgezeichnet. Aktuell nutzen Forschende in diesem Bereich unter anderem die strukturierte Beleuchtung, um eine erhöhte Auflösung zu erzielen. Dies ist momentan eines der am weitesten verbreiteten Verfahren, um dynamische Prozesse in lebenden Zellen darzustellen und abbilden zu können. Diese Methode erreicht eine Auflösung von 100 Nanometern mit hoher Bildrate, gleichzeitig werden die Proben beim Messen geschont. Diese Methode der hochauflösenden Fluoreszenzmikroskopie wird auch in der Arbeitsgruppe Biomolekulare Photonik an der Bielefelder Fakultät für Physik erfolgreich eingesetzt und weiterentwickelt, zum Beispiel um die Funktion der Leber oder Ausbreitungswege des HI Virus zu untersuchen.
Die mit dieser Methode aufgenommenen Rohbilder können allerdings nicht sofort von den Wissenschaftlern genutzt werden. „Die Mikroskopie-Methode erfordert eine sehr aufwändige, mathematische Bildrekonstruktion der aufgenommenen Daten. Erst hierdurch entsteht aus den im Mikroskop aufgenommenen Rohdaten ein hochauflösendes Bild“, erläutert Professor Dr. Thomas Huser, Leiter der Arbeitsgruppe Biomolekulare Photonik. Da dieser Schritt ein mathematisch komplexes Verfahren benutzt, das bisher nur wenigen Forschern zugänglich war, gab es bislang keine offene, für alle Forschenden einfach verfügbare Softwarelösung. Huser bewertet das als große Hürde, um die Technologie nutzen und weiterentwickeln zu können. Diese Lücke füllt nun die in Bielefeld entwickelte Software.
Dr. Marcel Müller aus der AG Biomolekulare Photonik ist es gelungen, eine solch universell einsetzbare Software zu erstellen. „Forschende weltweit arbeiten am Bau neuer, schnellerer und empfindlicherer Mikroskope zur strukturierten Beleuchtung, vor allem zur zweidimensionalen Abbildung lebender Zellen. Für die notwendige Nachverarbeitung müssen sie nun nicht mehr aufwendig eigene Lösungen entwickeln, sondern können direkt unsere Software nutzen, und dank der quelloffenen Verfügbarkeit auch auf ihre Probleme anpassen“, erklärt Müller. Die Software steht der weltweiten Forschungsgemeinschaft frei als „open source“-Lösung zur Verfügung und wurde schon kurz nach ihrer Ankündigung von Forschenden vor allem in Europa und Asien mehrfach angefordert und installiert. „Wir haben bereits viele positive Rückmeldungen bekommen“, sagt Marcel Müller. „Das spiegelt auch den Bedarf für diese Neuentwicklung wider.“
Originalveröffentlichung
M. Müller, V. Mönkemöller, S. Hennig, W. Hübner, and T. Huser; "Open source image reconstruction of super-resolution structured illumination microscopy data in ImageJ"; Nature Communications; 2016