Wie molekulare Fußbälle im Röntgenlaser zerplatzen
Studie zeigt Wirkung von Röntgenblitzen auf empfindliche Biomoleküle
Ein internationales Forschungsteam hat in Echtzeit verfolgt, wie Fußballmoleküle aus Kohlenstoff im Strahl eines Röntgenlasers zerplatzen. Die Untersuchung zeigt den zeitlichen Verlauf des Zerberstens, das weniger als eine billionstel Sekunde dauert, und hat Bedeutung für die Analyse empfindlicher Proteine und anderer Biomoleküle, die ebenfalls häufig mit Hilfe heller Röntgenlaserblitze durchleuchtet werden. Die Fußballmoleküle lösen sich langsamer und anders auf als erwartet, wie das Team um Nora Berrah von der Universität von Connecticut (US) und Robin Santra von DESY im Fachblatt „Nature Physics“ berichtet. Diese Beobachtung trägt zur verbesserten Analyse von Proteinen mit Röntgenlaserblitzen bei.
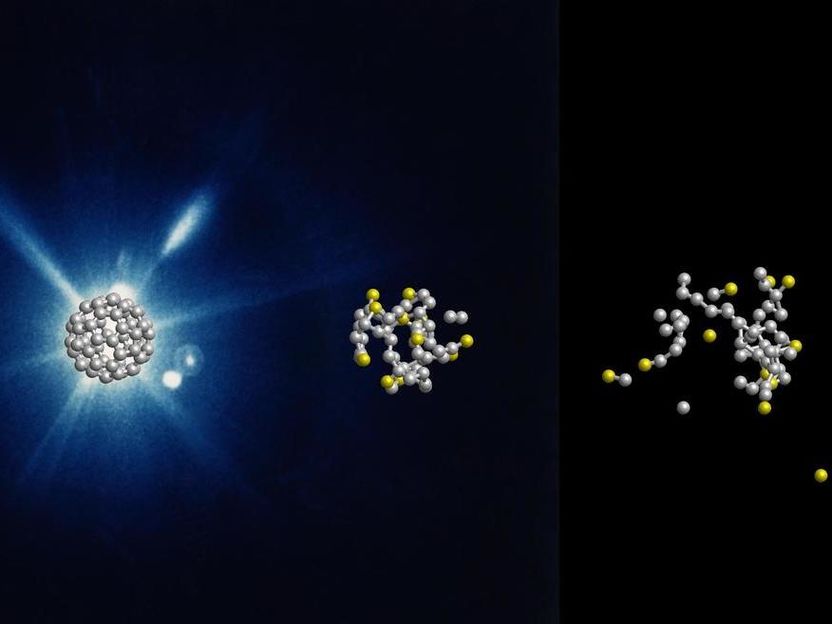
Computersimulation der Entwicklung eines Fußballmoleküls nach 0, 60 und 240 Femtosekunden.
DESY, Zoltan Jurek
Die Forscher hatten mit sogenannten Buckminster-Fullerenen experimentiert, kurz Buckyballs genannt. Diese kugelförmigen Moleküle bestehen aus 60 Kohlenstoffatomen, die in abwechselnden Fünf- und Sechsecken angeordnet sind wie die wabenförmigen Lederstücke eines Fußballs. „Buckyballs eignen sich gut als einfaches Modellsystem für Biomoleküle“, erläutert Santra, der Leitender Wissenschaftler bei DESY am Center for Free-Electron Laser Science (CFEL) und Physikprofessor an der Universität Hamburg ist. „Da sie nur aus einer Atomsorte bestehen und symmetrisch aufgebaut sind, lassen sie sich in Theorie und Experiment gut darstellen. Dies ist ein erster Schritt vor der Untersuchung von komplexeren Molekülen aus unterschiedlichen Atomsorten.“
Mit dem Röntgenlaser LCLS (Linac Coherent Light Source) am US-Forschungszentrum SLAC in Kalifornien beschossen die Wissenschaftler einzelne Fußballmoleküle mit jeweils nur etwa 20 Femtosekunden (billiardstel Sekunden) kurzen Blitzen und beobachteten deren Wirkung in Echtzeit mit einer zeitlichen Auflösung im Bereich von etwa zehn Femtosekunden. Die Daten zeigen, dass der Röntgenblitz aus etwa jedem fünften der 60 Kohlenstoffatome ein Elektron herausschlug. „Danach passiert zunächst einmal nichts. Erst nach einigen Dutzend Femtosekunden lösen sich nach und nach Kohlenstoffatome von dem Molekül“, berichtet Santra.
„Was dann folgt, ist keine Explosion“, erläutert der Physiker. „Stattdessen lösen sich die Buckyballs vergleichsweise langsam auf. Dabei dampfen nach und nach Kohlenstoffatome ab – und zwar viel mehr neutrale als elektrisch geladene, das war überraschend.“ Da die Fragmentation der Buckyballs auf dieser Zeitskala nicht explosionsartig erfolgt, sprechen die Forscher vom Abdampfen der Atome. Erst mit Hilfe der theoretischen Modellierung des Prozesses ließen sich die experimentellen Daten sinnvoll deuten.
„Typischerweise fliegen etwa 25 neutrale und nur 15 elektrisch geladene Kohlenstoffatome aus dem Molekül“, erläutert Santra. „Der Rest bildet Fragmente aus mehreren Atomen.“ Der ganze Prozess dauert rund 600 Femtosekunden. Das ist nach menschlichen Maßstäben immer noch unvorstellbar kurz, für die Strukturanalyse mit Röntgenlasern jedoch extrem lang. „In den typischerweise 20 Femtosekunden eines Röntgenlaserblitzes bewegen sich die Atome maximal 0,1 Nanometer – das ist im Bereich einzelner Atomdurchmesser und kleiner als die Messgenauigkeit der Strukturanalyse.“ Ein Nanometer ist ein millionstel Millimeter.
Für die Strukturanalyse von Proteinen züchten Forscher in der Regel kleine Kristalle aus den Biomolekülen. Der helle Röntgenlaserblitz wird dann am Kristallgitter gestreut und erzeugt ein typisches Beugungsmuster, aus dem sich die Kristallstruktur und damit auch die räumliche Struktur der einzelnen Proteine berechnen lässt. Die räumliche Struktur eines Proteins erlaubt Rückschlüsse auf seine genaue Funktionsweise. Die Proteinkristalle sind sehr empfindlich und verdampfen durch den Röntgenlaserblitz. Frühere Untersuchungen hatten jedoch gezeigt, dass der Kristall lange genug intakt bleibt, um vor dem Verdampfen das Beugungsbild zu erzeugen und damit seine räumliche Struktur preiszugeben.
Die neue Studie untermauert nun, dass dies auch bei einzelnen Molekülen der Fall ist, die sich nicht in einem Kristallgitter befinden. „Unsere Beobachtungen bei den Buckyballs spielen wahrscheinlich bei den meisten anderen Molekülen eine Rolle“, betont Santra. Da viele Biomoleküle sehr schwer zu kristallisieren sind, hoffen Forscher, künftig auch aus Ensembles nicht kristallisierter Proteine oder sogar bei einzelnen Biomolekülen mit dem Röntgenlaser die Struktur bestimmen zu können. Die jetzt erzielten Ergebnisse legten die Basis für ein tieferes Verständnis sowie eine quantitative Modellierung des Strahlenschadens in Biomolekülen durch Röntgenlaserblitze, schreiben die Wissenschaftler.
Originalveröffentlichung
"Femtosecond-resolved observation of the fragmentation of buckminsterfullerene following X-ray multiphoton ionization"; N. Berrah, A. Sanchez-Gonzalez, Z. Jurek, R. Obaid, H. Xiong, R. J. Squibb, T. Osipov, A. Lutman, L. Fang, T. Barillot, J. D. Bozek, J. Cryan, T. J. A. Wolf, D. Rolles, R. Coffee, K. Schnorr, S. Augustin, H. Fukuzawa, K. Motomura, N. Niebuhr, L. J. Frasinski, R. Feifel, C. P. Schulz, K. Toyota, S.-K. Son, K. Ueda, T. Pfeifer, J.P. Marangos and R. Santra; Nature Physics; 2019